1.引言
FISH(Fluorescence In Situ Hybridization)荧光原位杂交技术是一种在细胞或组织中定位特定核酸序列的重要方法。通过标记DNA或RNA探针,结合荧光显微镜,FISH技术能够在细胞水平上准确定位和检测特定的基因、染色体结构和功能区域,对于细胞遗传学、肿瘤学、发育生物学等领域的研究有着重要的应用价值。
2.FISH荧光原位杂交技术的原理
荧光原位杂交技术的原理是基于核酸互补配对的特性。当两条核酸单链片段存在适宜的条件下时,它们可以通过氢键结合形成稳定的双链分子,形成DNA-DNA、DNA-RNA或RNA-RNA双链结构。在荧光原位杂交中,探针是一种带有标记的DNA或RNA片段,这些标记可以是放射性同位素、荧光素、生物素、地高辛等非放射性物质。通过将荧光标记的DNA探针与待检测样品中的特定DNA序列结合,可以在细胞中定位和检测这些特定DNA序列的位置。[1][2]
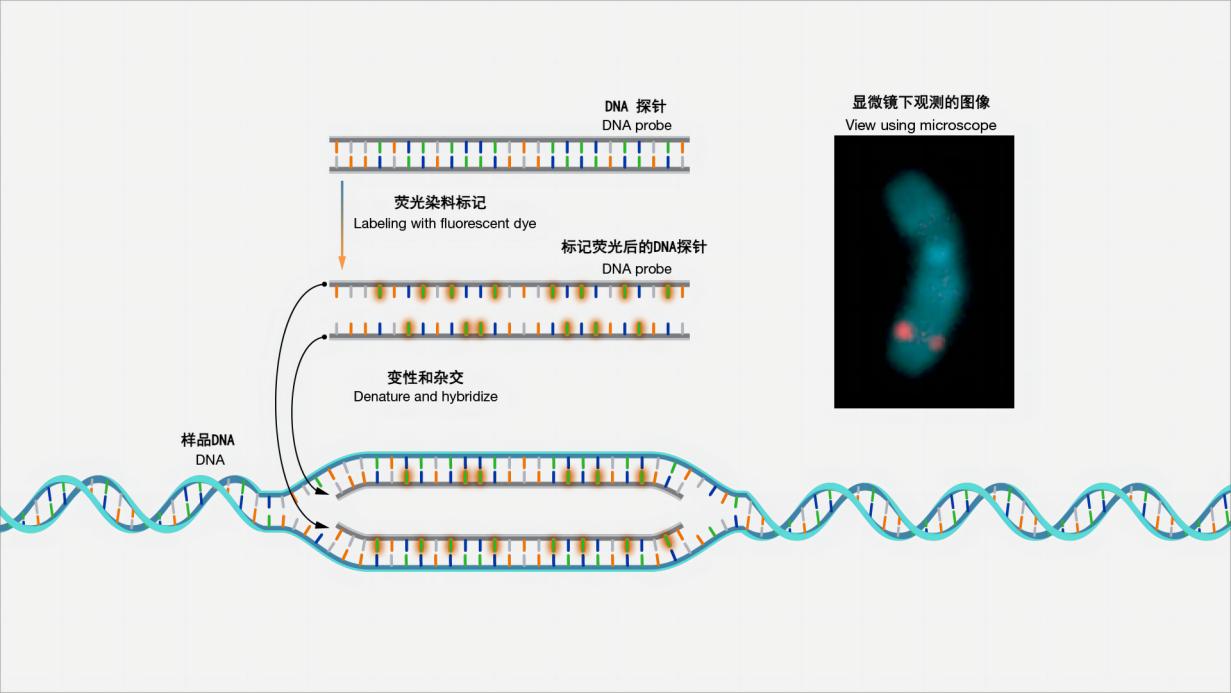
图 1 荧光原位杂交示意图
荧光原位杂交(简称FISH)是一种实验室技术,用于检测和定位染色体上的特定DNA序列。在这种技术中,来自个体的完整染色体组被固定在玻璃载玻片上,然后暴露于“探针”—— 一小段带有荧光染料标记的纯化DNA。荧光标记的探针会找到并与染色体组中的相配序列结合。借助特殊的显微镜,可以看到荧光探针结合的染色体和亚染色体位置。(图片与解释来源:Fluorescence In Situ Hybridization (FISH) (genome.gov))
荧光原位杂交技术最初是为了在哺乳动物染色体中检测特定DNA序列而发展的,后来也被应用于植物染色体的研究。利用荧光标记的DNA探针与待检测样品中的特定DNA序列结合,可以在细胞中形成荧光信号,从而使特定DNA序列在细胞中可见。常用的检测方法是荧光显微镜下的荧光信号检测,在光学或电子显微镜下观察和定位目标核酸序列的位置,从而对细胞或组织中的基因表达进行定量和定位分析。这项技术有助于研究染色体的结构和功能,以及检测染色体异常和遗传变异。[1][3]
这样的技术提供了研究者一个有力的工具,能够深入探索细胞和组织中的基因组信息。通过观察和定位特定的DNA序列,研究者可以更好地理解基因表达调控、染色体结构和功能等方面的机制。同时,荧光原位杂交技术也在医学诊断和遗传学研究中发挥着重要作用,例如,可以用于检测肿瘤细胞中的染色体异常或基因突变,为疾病的诊断和治疗提供重要依据。
3.FISH技术应用案例
FISH 是乳腺癌中公认的HER2基因状态评估方法。乳腺癌 HER2 状态的检测是筛选接受抗 HER2靶向治疗(曲妥珠单抗和拉帕替尼等)患者的前提条件。运用FISH技术评价乳腺癌HER2状态被认为是为早期或晚期乳腺癌患者制定合理系统治疗策略的金标准。参照免疫组化检测结果,医生确定潜在扩增的浸润性肿瘤组织细胞,再使用荧光显微镜观察并统计细胞核中红色 HER2 与绿色CEP18(chromosome 17 centromere)荧光信号的数目。根据2018版美国临床肿瘤学会/美国病理医师学院(ASCO/CAP)乳腺癌HER2检测指南,临床上要求至少对20个边界完整的癌细胞核内信号进行定量统计,依据NHER2/NCEP17 比值(其中N 为数量)和HER2平均拷贝数(CHER2)这两项指标进行分级。当 NHER2/NCEP17≥2.0,且CHER2/Ncell≥4.0时,此种情况判为FISH阳性,其中Ncell为细胞数量。[6][8]
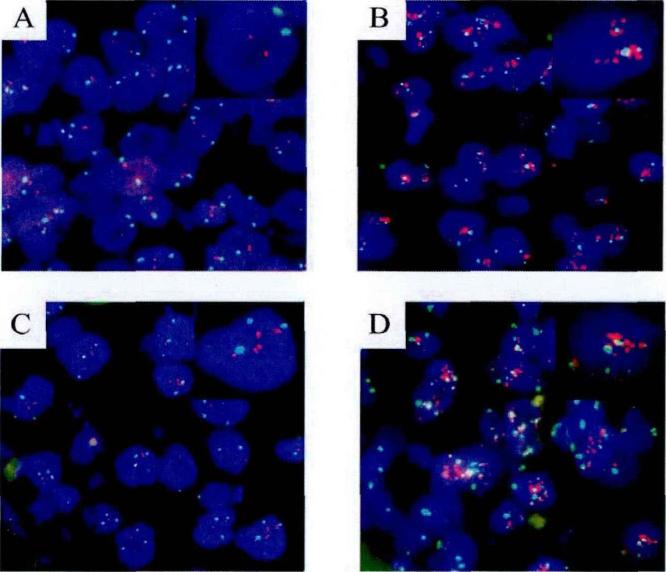
图 2 普通荧光显微镜检测GER2基因扩增状态[8]
HER2基因状态通过FISH确定。A,HER2基因未扩增,没有多体性17。B,HER2基因扩增,带有多体性17。C,HER2基因未扩增,没有多体性17。D,HER2基因扩增,带有多体性。(图片与解释来源:马丽.中国乳腺癌HER2基因检测、临床病理特征分析及流行病学研究[D].北京协和医学院,2012.)
4.成像解决方案对比
| 观察方式 |
优点 |
缺点 |
| 宽场荧光显微镜 |
- 用于FISH病理诊断的主要成像工具之一
- 相对较简单易用,成本较低
- 可以在细胞中较容易确定FISH点坐标
|
- 受到光学衍射极限的限制,成像分辨率较低
- 无法准确计数核内距离小于分辨率的荧光探针。
- 不适用于成像组织切片
|
| 激光扫描共聚焦显微镜 |
- 提高了成像信噪比,相比宽场荧光显微镜具有更好的成像质量
- 可以通过荧光免疫组织化学同时可视化多个突触结构标记
|
- 成像速度较慢,不适合实际的FISH诊断应用
- 使用光电倍增管的量子效率相对较低
|
| 转盘共聚焦显微镜 |
- 具有更快的成像速度和较高的成像分辨率
- 对于同时量化多个突触组分的数量和估算组织切片中蛋白质水平是出色的工具。
- 更快更好的三维成像
- CCD相机具有较高的量子效率,成像质量较高。
- 减少光漂白和光毒性
|
|
详细介绍:
宽场荧光显微镜和荧光病理切片扫描仪是目前进行FISH病理诊断的主要成像工具。然而,宽场荧光显微镜由于受到光学衍射极限的限制,当采用波长为561 nm 的激发光时,FISH 探针的成像分辨率约为300 nm,当细胞核中两个或多个的荧光探针距离小于成像分辨率时,将无法对其进行准确计数。要实现对阳性患者的HER2基因水平的精确统计,需要采用更高分辨率的成像技术。另外,宽场荧光显微镜不适用于成像组织切片,因为会检测到太多的离焦模糊。例如 FISH 染色的染色体区域。虽然在宽场显微镜中可以很容易地确定FISH点的z坐标,染色体边界过于模糊和不清晰。宽视场显微镜还无法根据复染图像精确确定 3D 细胞核边界,因为细胞核图像的顶部和底部存在大量失焦雾霾。[5][6]
激光扫描共聚焦显微成像技术(CLSM)利用物理针孔将离焦信号滤除,相比宽场荧光显微镜,明显地提高了成像信噪比。Ram等使用共聚焦荧光显微镜定位染色体目标的DNA片段并定量计算空间距离,提出了一种用于分割和检测 3D FISH斑点的算法。[6] CLSM的出现显著提高了通过荧光免疫组织化学同时可视化多个突触结构标记的能力。同时评估多个标记物的优势在于可以确定不同抗原共定位的频率。此外,使用荧光可以确定已鉴定结构内的相对抗原浓度。虽然CLSM是用于确定标记的突触组分密度的优秀研究工具,但并不一定是量化标记抗原的最佳工具。由于CLSM采用点扫描方式进行成像,成像速度较慢,目前在实际的FISH诊断中没有广泛应用。虽然理想情况下应该使用宽场荧光显微镜进行定量荧光测量(Murray等人,2007年;Swedlow等人,2002年),但宽场荧光显微镜不适用于成像组织切片,因为会检测到太多的离焦模糊。介于CLSM和常规显微镜之间,转盘共聚焦显微镜收集的信息比CLSM多10倍(Sandison和Webb,1994年)。
转盘共聚焦显微镜对于同时量化多个突触组分的数量以及估算组织切片中突触结构中蛋白质水平的工具是异常出色的。此外,转盘共聚焦显微镜中用于图像检测的CCD相机具有较高的量子效率(通常超过90%),而大多数CLSM上使用的光电倍增管的量子效率相对较低(约为25%)(Murray等人,2007年;Sandison和Webb,1994年;Shaw,2006年;Wang等人,2005年)。使用转盘共聚焦收集3D图像堆栈的过程比使用CLSM收集速度快得多。提高的速度减少了在焦平面上下发生的光漂白量,这种效应在成像厚组织切片时会放大,因为会在大量的焦平面进行成像。此外,速度的增加也有助于收集大多数立体学采样方案所需的大数据集。[7]
5.瑆科的成像解决方案和成像结果
在基于不影响数据质量的同时提高实验室工作效率的愿景之上,我司开发了 SpinDisk 系列,该系列提供了实现快速柔和共聚焦成像最优惠的解决方案。

图 3 瑆科转盘共聚焦显微系统-SpinDisk Basic
瑆科转盘共聚焦显微镜SpinDisk Basic是一种先进的显微镜成像技术,通过在样品和探测器之间引入光盘,能够实现在不同焦平面上的快速成像,提高成像深度和分辨率。与传统的荧光显微镜相比,转盘共聚焦显微镜具有更高的成像速度和分辨率,能够实现更准确的三维成像,同时减少光淬灭和荧光信号衰减对图像质量的影响。
FISH(荧光原位杂交)技术在观察过程中可能面临的挑战主要包括:
- 荧光信号弱:荧光信号可能受到多种因素的影响,如探针的亲和性、样品的处理方式等,导致信号弱,难以观察和分析。
- 背景噪声干扰:样品制备过程中可能存在背景噪声,例如非特异性结合或自发发光等,与目标信号混合,干扰图像的清晰度和准确性。
- 多通道成像叠加:在使用多个荧光通道时,不同通道下的信号可能会相互叠加,使得分析和识别特定信号变得困难。
针对这些挑战,瑆科转盘共聚焦显微系统SpinDisk Basic提供了一系列解决方案:
- 高亮度激光光源:系统采用多通道连续多模激光器(四合一激光器)作为光源,提供高亮度的激光,以增强荧光信号的强度,从而使得信号更容易被观察和分析。 四合一激光系统的优点在于它能够提供多波长的激光光源,能够同时激发多种荧光染料或标记物,在一次实验中获得更全面的信息。这对于细胞和组织的多色荧光成像、共定位和共表达研究具有重要意义。
- 多通道激发一键切换:系统配置了电动滤光轮。通过软件控制电动滤光轮与多通道激光器,实现了多通道激发的一键切换功能,可以快速调整激发和发射通道,避免不同通道下的信号叠加问题。
- 优质滤光片和背景抑制技术:系统配备了优质的滤光片,能够有效滤除背景噪声,并采用瑆科开发的转盘配套软件,背景抑制技术,如智能背景校正算法等,以提高图像的清晰度和信噪比。
- 高灵敏度探测器:系统采用高灵敏度的探测器,BSI sCOM相机,能够有效地捕获荧光信号,并提供高质量的成像结果。
将SpinDisk Basic与FISH技术相结合,通过这些解决方案的应用,可以克服传统成像方案的一些局限性,实现更高质量的FISH成像的同时具有丰富的功能,包括但不限于:
- 大图像拼接:系统具有大图像拼接功能,可以将多个局部图像拼接成一个大图像,从而扩展成像范围,获得更大的视野。
- 实时预览存图:系统提供实时图像预览功能,使用户能够实时观察样品的荧光信号并进行图像采集。同时,用户可以将所观察到的图像直接存储下来,方便后续分析和处理。
- 多通道成像与融合:系统支持多通道荧光成像,可以同时捕获多个荧光通道的图像。这使得用户可以在同一样品上同时检测多个目标DNA序列或标记物,从而获得更加全面的信息。此外,系统还提供图像融合功能,将不同通道的图像叠加在一起,以便于观察和分析。
- 图像处理:系统配备了图像处理软件,可以进行图像的调整、增强和修正,以提高图像质量和对比度,帮助用户更清晰地观察样品结构和信号。
- 三维重构:系统支持对成像样品进行三维重构,即从一系列图像中重建出三维样品结构。这使得用户可以在整个样品体积内观察目标DNA序列的分布和位置,从而更全面地了解样品的空间结构。
- 多色荧光定位处理:系统可以针对多个不同的荧光标记进行定位处理,即确定不同荧光信号的位置和分布情况。这对于同时检测多个目标或标记物的FISH实验非常重要。
- Z-stack数据处理:系统支持对Z-stack数据进行处理,即沿着Z轴方向获取一系列图像,然后将它们叠加起来以重建样品的三维结构。这有助于用户更全面地了解样品的立体形态和空间分布。
- 图像分析:系统提供图像分析工具,可以对图像进行定量分析和测量,例如计算荧光信号的强度、分布和关联性等,从而帮助用户量化分析实验结果。
- 成像数据管理:系统具有成像数据管理功能,可以对采集的图像和数据进行存储、管理和检索,确保数据的安全性和完整性,并方便用户后续的数据处理和回顾。
- 3D成像渲染:系统支持对三维重构后的样品进行渲染和显示,可以实现多种不同的三维成像效果,以便于用户更直观地观察和理解样品的立体结构。
- 硬件控制:系统具有完善的硬件控制功能,可以精确控制光源、滤光片、镜头等硬件参数,以保证成像的稳定性和准确性。
- 多维度自动扫描:系统支持多维度自动扫描,可以根据用户设定的扫描参数自动在多个维度上对样品进行扫描,从而实现更全面的成像覆盖和更高效的数据采集。
SpinDisk Basic成像展示:
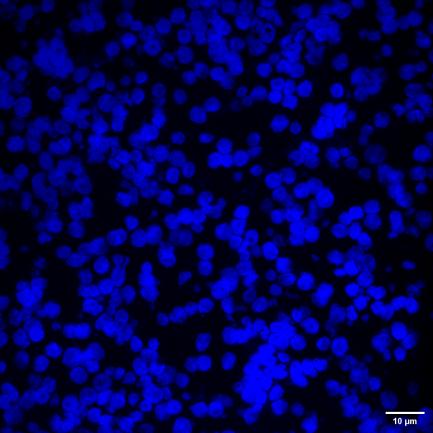


图 4 405/488/525三个通道60X 1.2NA 宫颈癌细胞FISH探针样本采图
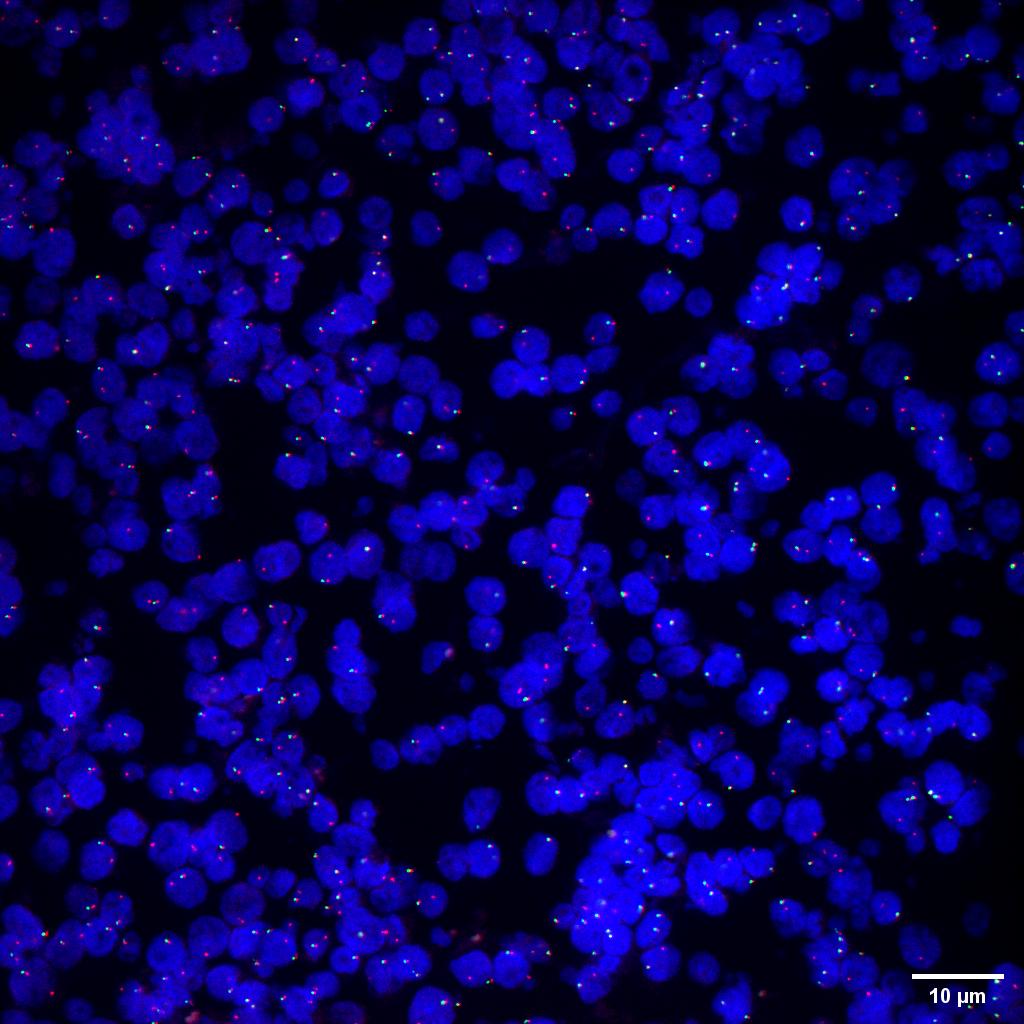
图 5 SpinDisk Basic-60X 1.2NA-405/488/525-宫颈癌细胞FISH探针样本多色融合
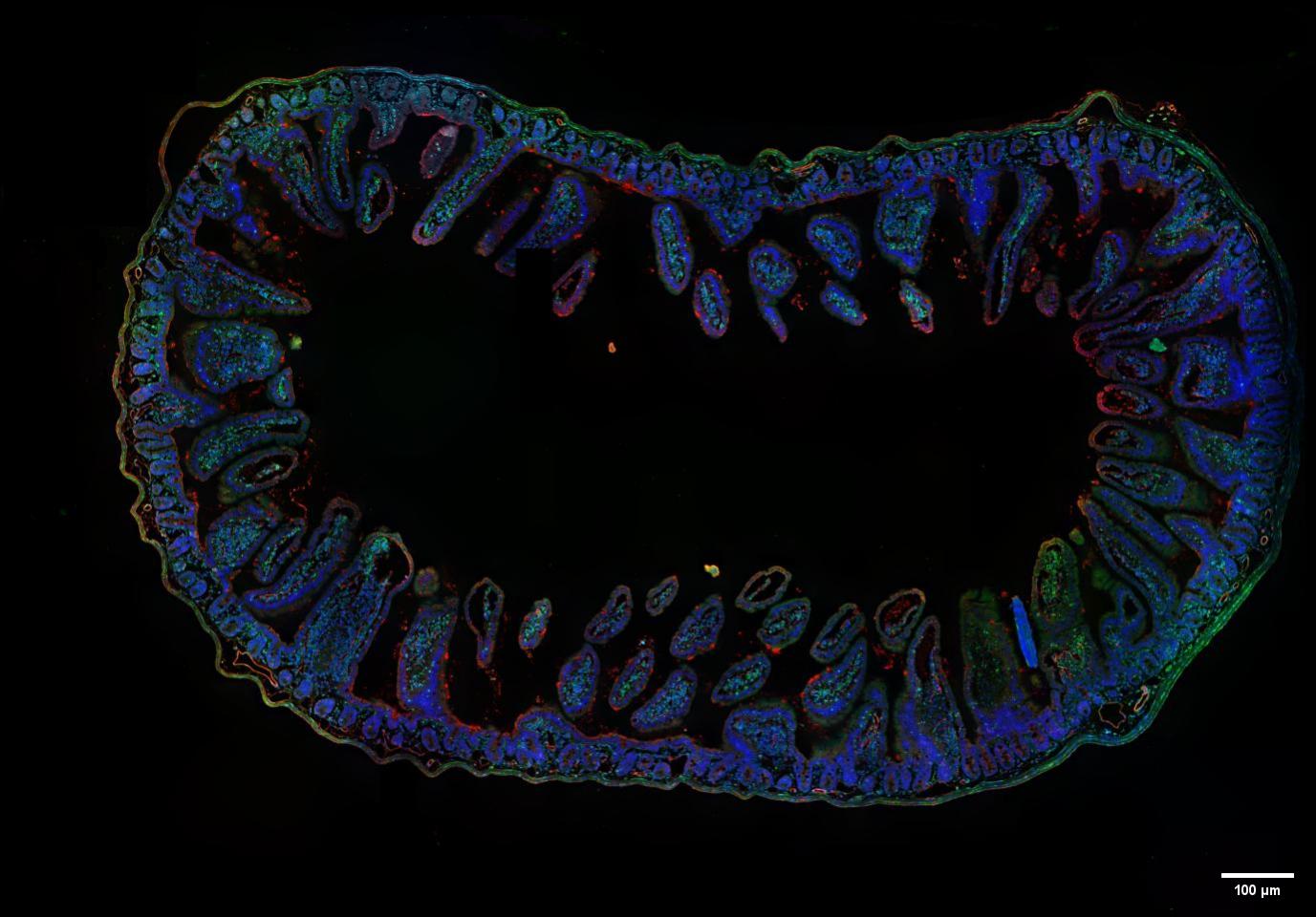
图 6 SpinDisk Basic-20X 0.5NA-405/488/525-小鼠肠组织包埋切片样本多色融合与大图拼接-全图尺寸1.8mmX1.25mm由56张局部图拼接而成
SpinDisk Basic 转盘共聚焦系统结合20X 0.5NA物镜和多通道激光(405nm、488nm和525nm),提供了多色融合和大图拼接的功能,为用户提供了更全面、更清晰的成像解决方案。
利用SpinDisk Basic的转盘共聚焦模式,我们可以快速捕获不同通道的荧光图像,从而获得目标样品在不同波长下的荧光信号。通过优化的图像处理算法,这些多通道的图像可以被准确地融合在一起,形成彩色的合成图像,展现出样品的多色荧光标记和分布情况。
此外,SpinDisk Basic还具有强大的大图拼接功能,可以将多个局部图像拼接成一个大图像,扩展了成像范围,提高了成像分辨率和视野。这为用户提供了更全面、更详细的样品信息,帮助他们更好地理解和分析样品的结构和特征。在拼接图像过程中,我们采用了优化的算法,确保图像的连续性和准确性。
公司介绍:
瑆科仪器于2019在新加坡成立,拥有优秀的研发团队,核心成员具备数十年光学科技与行业背景,专注于显微镜技术的创新与应用,并先后与新加坡南洋理工大学(NTU),新加坡国立大学(NUS)和A-Star联合开发高端显微镜系统。我们专注于共聚焦,超分辨显微等相关领域,致力于生产科研与工业级的高端显微镜产品。 我们的核心产品是高分辨显微镜系列,采用优秀的光学技术和图像处理算法,具有卓越的成像性能,以满足客户在高分辨、快速成像等方面的需求。
6.引文
[1]Shakoori AR. Fluorescence In Situ Hybridization (FISH) and Its Applications. Chromosome Structure and Aberrations. 2017 Feb 10:343–67. doi: 10.1007/978-81-322-3673-3_16. PMCID: PMC7122835.
[2]Jin, L. and Lloyd, R.V. (1997), In situ hybridization: Methods and applications. J. Clin. Lab. Anal., 11: 2-9. https://doi.org/10.1002/(SICI)1098-2825(1997)11:1<2::AID-JCLA2>3.0.CO;2-F
[3]刘玉玲,刘林杰,彭仁海.荧光原位杂交技术的发展及其在植物基因组研究中的应用[J].分子植物育种,2018,16(17):5696-5703.DOI:10.13271/j.mpb.016.005696.
[4]Fluorescence In Situ Hybridization (FISH)By: Clare O'Connor, Ph.D. (Biology Department, Boston College) © 2008 Nature Education Citation: O'Connor, C. (2008) Fluorescence in situ hybridization (FISH). Nature Education 1(1):171
[5]Kozubek, M., Kozubek, S., Lukášová, E., Bártová, E., Skalníková, M., Matula, P., Matula, P., Jirsová, P., Cafourková, A. and Koutná, I. (2001), Combined confocal and wide-field high-resolution cytometry of fluorescent in situ hybridization-stained cells. Cytometry, 45: 1-12.
[6]吴寅,梁永,张洁,et al.荧光原位杂交扩增探针的结构光照明超分辨成像与计数[J].激光与光电子学进展, 2024, 61(4):0411009.DOI:10.3788/LOP231182.
[7]Kenneth N. Fish, Robert A. Sweet, Anthony J. Deo, David A. Lewis,An automated segmentation methodology for quantifying immunoreactive puncta number and fluorescence intensity in tissue sections,Bran Research,Volume 1240,2008,Pages 62-72,ISSN 0006-8993
[8]马丽.中国乳腺癌HER2基因检测、临床病理特征分析及流行病学研究[D].北京协和医学院,2012.